Exam: Rozdiel medzi revíziami
(→Pokyny ku skúške) |
|||
| Riadok 1: | Riadok 1: | ||
| − | == | + | == Exam rules == |
| − | + | The main part is '''written''': | |
| − | * | + | * You need at least 50% of points |
| − | * | + | * Time 3 hours |
| − | * | + | * About 50% of points for simple questions, |
| − | * | + | ** examples on this page |
| − | * | + | ** in case of interest tutorial session before exam |
| + | * The rest of the questions mostly designing/modifying an algorithm or model | ||
| + | * Online or in person, depending on circumstances | ||
| + | * You can use pen, simple calculator and a cheat sheet up to 2 A4 two-sided sheets | ||
| − | + | ===Written exam, online version=== | |
| − | * | + | * Exam questions and submission in Moodle (e-mail for guests) |
| − | + | * MS teams: annoucements, questions | |
| − | * | + | * Write in an editor, create pdf or write on paper, scan/photo, convert to pdf |
| − | * | + | * Allowed aids: |
| − | * | + | ** Same as in person (incl. cheat sheet) |
| − | * | + | ** Plus: Text and image editors, software for digitization of handwritten pages, MS Teams to communicate with instructors Moodle for getting and submitting exam |
| − | * MS Teams | + | * Not allowed: |
| − | * | + | ** Communication with other persons except instructors |
| + | ** Other webpages | ||
| + | ** Other software (e.g. specialized bioinformatics programs, compilers) | ||
| − | + | ===Oral exam=== | |
| − | * | + | * Only for online exam |
| − | * | + | * Videocall in MS Teams |
| − | * | + | * After written exam, time slots over several days |
| + | * We will discuss your exam | ||
| + | * You should be able to explain your answers in detail | ||
| + | * Oral exam influences exam grade | ||
| + | * If you are unable to explain your answers, you will get Fx | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | ===“Second chance” exam=== | |
| + | * The same for as the first or oral-only | ||
| + | * The dates arranged with those who need them | ||
<!-- | <!-- | ||
| Riadok 37: | Riadok 41: | ||
--> | --> | ||
| − | ==Sylabus | + | ==Sylabus and examples of problem== |
| − | + | Below we list the most important concepts that both biologists and computer scientists should know form this course. | |
| − | + | We also list simple questions. Questions of this type will comprise approximately 50% of the exam. Not all of these questions will be used on the exam and particular string, numbers and sequences will differ. | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | === | + | ===Sequencing and genome assembly=== |
| + | |||
| + | DNA sequencing and its use, sequencing read, paired reads, contigs, shortest common superstring problem, de Bruijn graphs | ||
| + | |||
Sekvenovanie DNA a jeho využitie, čítanie (read), spárované čítania, kontig, problém najkratšieho spoločného nadslova, de Bruijnove grafy | Sekvenovanie DNA a jeho využitie, čítanie (read), spárované čítania, kontig, problém najkratšieho spoločného nadslova, de Bruijnove grafy | ||
| − | * | + | * Find the shortest common superstring of strings GACAATAA, ATAACAC, GTATA, TAATTGTA. |
| − | * | + | * Find the de Bruijn graph for k=2 (nodes will be pairs of nucleotides) and reads CCTGCC, GCCAAC |
| + | |||
| + | ===Sequence alignment=== | ||
| + | |||
| + | The problem of local and global alignment of two sequences, dynamic programming algorithms, scoring matrix and its probabilistic meaning, statistical significance (E-value, P-value), heuristic search of local alignments (BLAST), whole-genome and multiple alignments | ||
| − | |||
Problém lokálneho a globálneho zarovnania dvoch sekvencií, jeho riešenie pomocou dynamického programovania, skórovacia matica a jej pravdepodobnostný význam, štatistická významnosť (E-value, P-value), heuristické hľadanie lokálnych zarovnaní (BLAST), celogenómové a viacnásobné zarovnania | Problém lokálneho a globálneho zarovnania dvoch sekvencií, jeho riešenie pomocou dynamického programovania, skórovacia matica a jej pravdepodobnostný význam, štatistická významnosť (E-value, P-value), heuristické hľadanie lokálnych zarovnaní (BLAST), celogenómové a viacnásobné zarovnania | ||
| − | * | + | * Fill in the dynamic programing matrices for local and global alignment of sequences TACGT a CAGGATT, where the match has score +3, mismatch -1, gap -2. Reconstruct also the optimal alignments found by the dynamic programming algorithm |
| − | * | + | |
| + | * Compute the score of the alignment shown below using the scoring matrix shown below, gap opening penalty -5, gap extension penalty -2 for each additional base. Find a global alignment with a higher score for these two sequences and compute its score. (It is not necessary to find the optimal alignment; you can use any method to arrive at the answer.) | ||
| + | |||
<pre> | <pre> | ||
| − | + | Alignment: Matrix: | |
ATAGTTTAA A C G T | ATAGTTTAA A C G T | ||
A-GGG--AA A 2 -2 -1 -2 | A-GGG--AA A 2 -2 -1 -2 | ||
| Riadok 68: | Riadok 76: | ||
</pre> | </pre> | ||
| − | * | + | * Consider BLASTn algorithm starting from seeds of size w=3. How many seeds it finds while comparing sequences GATTACGGAT and CAGGATT? List all found seeds. |
| + | |||
| + | ===Gene finding=== | ||
| + | |||
| + | Gene, exon, intron, mRNA, splicing and alternative splicing, genetic code, hidden markov model (HMM), its states, transition and emission probabilities, use of HMMs in gene finding | ||
| − | |||
Gén, exón, intrón, mRNA, zostrih a alternatívny zostrih, kodón, genetický kód, skrytý Markovov model (HMM), jeho stavy, pravdepodobnosti prechodu a emisie, použitie HMM na hľadanie génov | Gén, exón, intrón, mRNA, zostrih a alternatívny zostrih, kodón, genetický kód, skrytý Markovov model (HMM), jeho stavy, pravdepodobnosti prechodu a emisie, použitie HMM na hľadanie génov | ||
| − | |||
| − | === | + | * What is the probablity of generating sequence AGT using sequence of states 1,2,1 in the HMM below? |
| + | <pre> | ||
| + | The HMM has three states 1, 2, 3. It always starts in state 1. | ||
| + | Transition probabilities: | ||
| + | From 1 to 1: 0.99 | ||
| + | From 1 to 2: 0.01 | ||
| + | From 2 to itself: 0.9 | ||
| + | From 2 to 1: 0.05 | ||
| + | From 2 to 3: 0.05 | ||
| + | From 3 to itself: 0.99 | ||
| + | From 3 to 2: 0.01 | ||
| + | Emmision probabilities in state 1: | ||
| + | A 0.25, C 0.25, G 0.25, T 0.25 | ||
| + | Emmision probabilities in state 2: | ||
| + | A 0.3, C 0.2, G 0.2, T 0.3 | ||
| + | Emmision probabilities in state 3: | ||
| + | A 0.2, C 0.4, G 0.3, T 0.1 | ||
| + | </pre> | ||
| + | |||
| + | ===Evolution and comparative genomics=== | ||
| + | |||
Fylogenetický strom (zakorenenený a nezakorenený), metóda maximálnej úspornosti (parsimony), metóda spájania susedov (neighbor joining), metóda maximálnej vierohodnosti (maximum likelihood), Jukes-Cantorov model substitúcií a zložitejšie substitučné matice, homológ, paralóg, ortológ, detekcia pozitívneho a negatívneho výberu, fylogenetické HMM, likelihood ratio test | Fylogenetický strom (zakorenenený a nezakorenený), metóda maximálnej úspornosti (parsimony), metóda spájania susedov (neighbor joining), metóda maximálnej vierohodnosti (maximum likelihood), Jukes-Cantorov model substitúcií a zložitejšie substitučné matice, homológ, paralóg, ortológ, detekcia pozitívneho a negatívneho výberu, fylogenetické HMM, likelihood ratio test | ||
Verzia zo dňa a času 10:52, 16. december 2021
Obsah
- 1 Exam rules
- 2 Sylabus and examples of problem
- 2.1 Sequencing and genome assembly
- 2.2 Sequence alignment
- 2.3 Gene finding
- 2.4 Evolution and comparative genomics
- 2.5 Expresia génov, regulácia, motívy
- 2.6 Proteíny
- 2.7 RNA
- 2.8 Populačná genetika
- 2.9 Ďalšie dôležité znalosti z cvičení pre informatikov
- 2.10 Ďalšie dôležité znalosti z cvičení pre biológov
Exam rules
The main part is written:
- You need at least 50% of points
- Time 3 hours
- About 50% of points for simple questions,
- examples on this page
- in case of interest tutorial session before exam
- The rest of the questions mostly designing/modifying an algorithm or model
- Online or in person, depending on circumstances
- You can use pen, simple calculator and a cheat sheet up to 2 A4 two-sided sheets
Written exam, online version
- Exam questions and submission in Moodle (e-mail for guests)
- MS teams: annoucements, questions
- Write in an editor, create pdf or write on paper, scan/photo, convert to pdf
- Allowed aids:
- Same as in person (incl. cheat sheet)
- Plus: Text and image editors, software for digitization of handwritten pages, MS Teams to communicate with instructors Moodle for getting and submitting exam
- Not allowed:
- Communication with other persons except instructors
- Other webpages
- Other software (e.g. specialized bioinformatics programs, compilers)
Oral exam
- Only for online exam
- Videocall in MS Teams
- After written exam, time slots over several days
- We will discuss your exam
- You should be able to explain your answers in detail
- Oral exam influences exam grade
- If you are unable to explain your answers, you will get Fx
“Second chance” exam
- The same for as the first or oral-only
- The dates arranged with those who need them
Sylabus and examples of problem
Below we list the most important concepts that both biologists and computer scientists should know form this course.
We also list simple questions. Questions of this type will comprise approximately 50% of the exam. Not all of these questions will be used on the exam and particular string, numbers and sequences will differ.
Sequencing and genome assembly
DNA sequencing and its use, sequencing read, paired reads, contigs, shortest common superstring problem, de Bruijn graphs
Sekvenovanie DNA a jeho využitie, čítanie (read), spárované čítania, kontig, problém najkratšieho spoločného nadslova, de Bruijnove grafy
- Find the shortest common superstring of strings GACAATAA, ATAACAC, GTATA, TAATTGTA.
- Find the de Bruijn graph for k=2 (nodes will be pairs of nucleotides) and reads CCTGCC, GCCAAC
Sequence alignment
The problem of local and global alignment of two sequences, dynamic programming algorithms, scoring matrix and its probabilistic meaning, statistical significance (E-value, P-value), heuristic search of local alignments (BLAST), whole-genome and multiple alignments
Problém lokálneho a globálneho zarovnania dvoch sekvencií, jeho riešenie pomocou dynamického programovania, skórovacia matica a jej pravdepodobnostný význam, štatistická významnosť (E-value, P-value), heuristické hľadanie lokálnych zarovnaní (BLAST), celogenómové a viacnásobné zarovnania
- Fill in the dynamic programing matrices for local and global alignment of sequences TACGT a CAGGATT, where the match has score +3, mismatch -1, gap -2. Reconstruct also the optimal alignments found by the dynamic programming algorithm
- Compute the score of the alignment shown below using the scoring matrix shown below, gap opening penalty -5, gap extension penalty -2 for each additional base. Find a global alignment with a higher score for these two sequences and compute its score. (It is not necessary to find the optimal alignment; you can use any method to arrive at the answer.)
Alignment: Matrix:
ATAGTTTAA A C G T
A-GGG--AA A 2 -2 -1 -2
C -2 1 -2 -1
G -1 -2 1 -2
T -2 -1 -2 2
- Consider BLASTn algorithm starting from seeds of size w=3. How many seeds it finds while comparing sequences GATTACGGAT and CAGGATT? List all found seeds.
Gene finding
Gene, exon, intron, mRNA, splicing and alternative splicing, genetic code, hidden markov model (HMM), its states, transition and emission probabilities, use of HMMs in gene finding
Gén, exón, intrón, mRNA, zostrih a alternatívny zostrih, kodón, genetický kód, skrytý Markovov model (HMM), jeho stavy, pravdepodobnosti prechodu a emisie, použitie HMM na hľadanie génov
- What is the probablity of generating sequence AGT using sequence of states 1,2,1 in the HMM below?
The HMM has three states 1, 2, 3. It always starts in state 1. Transition probabilities: From 1 to 1: 0.99 From 1 to 2: 0.01 From 2 to itself: 0.9 From 2 to 1: 0.05 From 2 to 3: 0.05 From 3 to itself: 0.99 From 3 to 2: 0.01 Emmision probabilities in state 1: A 0.25, C 0.25, G 0.25, T 0.25 Emmision probabilities in state 2: A 0.3, C 0.2, G 0.2, T 0.3 Emmision probabilities in state 3: A 0.2, C 0.4, G 0.3, T 0.1
Evolution and comparative genomics
Fylogenetický strom (zakorenenený a nezakorenený), metóda maximálnej úspornosti (parsimony), metóda spájania susedov (neighbor joining), metóda maximálnej vierohodnosti (maximum likelihood), Jukes-Cantorov model substitúcií a zložitejšie substitučné matice, homológ, paralóg, ortológ, detekcia pozitívneho a negatívneho výberu, fylogenetické HMM, likelihood ratio test
- Na strome nižšie nájdite najúspornejšie ancestrálne znaky pre stĺpec zarovnania TTAAA (v poradí glum, hobit, človek, elf, ork). Odpoveď môžete spočítať ľubovoľným spôsobom.
Glum ----|
|----|
Hobit ----| |----|
| |
Človek ---------| |
|---
Elf --------| |
|--------|
Ork --------|
- Nájdite najúspornejší strom pre zarovnanie uvedené nižšie. Aká je jeho cena (koľko mutácií je nutných na vysvetlenie týchto sekvencií)? Odpoveď môžete spočítať ľubovoľným spôsobom.
vtáčik biely ACAACGTCT vtáčik čierny TCTGAATCA vtáčik sivý TGTGAAAGA vtáčik modrý ACTACGTCT vtáčik zelený TGTGAAAGA
- Uvažujme maticu vzdialeností uvedenú nižšie. Ktorú dvojicu vrcholov spojí metóda spájania susedov ako prvú a aká bude nová matica po spojení?
biely čierny sivý modrý
vtáčik biely 0 5 7 4
vtáčik čierny 5 0 8 5
vtáčik sivý 7 8 0 5
vtáčik modrý 4 5 5 0
- Uvažujme strom pre gluma, hobita atď uvedený v príklade vyššie (bol by v zadaní), pričom každá hrana má rovnakú dĺžku a pravdepodobnosť každej mutácie na jednej hrane je 0.1 (t.j. napr. Pr(C|A,t)=0.1) a teda pravdepodobnosť zachovania tej istej bázy je 0.7, pravdepodobnosť každej bázy v koreni je 0.25. Aká je pravdepodobnosť, že v listoch dostaneme TTAAA a vo vnútorných vrcholoch samé Áčka? Nájdite priradenie ancestrálnych báz vo vnútorných vrcholoch, ktoré má väčšiu pravdepodobnosť a spočítajte, aká tá pravdepodobnosť je (nemusíte nájsť najlepšie možné priradenie).
Expresia génov, regulácia, motívy
Určovanie génovej expresie pomocou microarray alebo sekvenovaním RNA-seq, hierarchické zhlukovanie, klasifikácia, reprezentácia sekvenčných motívov (väzobné miesta transkripčných faktorov) ako konsenzus, regulárny výraz a PSSM, hľadanie nových motívov v sekvenciách, consensus pattern problem, hľadanie motívu pomocou pravdepodobnostných modelov (EM algoritmus)
- Uvažujme microarray experimenty pre 5 génov. Medzi každými dvomi génmi sme spočítali vzdialenosť ich profilov expresie a dostali sme tabuľku vzdialeností uvedenú nižšie. Nájdite hierarchické zhlukovanie týchto génov, pričom vzdialenosť medzi dvoma zhlukmi (clustrami) bude vzdialenosť najbližších génov v nich. Uveďte aj v akom poradí ste jednotlivé zhluky tvorili.
A B C D E
gén A 0 0.6 0.1 0.3 0.7
gén B 0.6 0 0.5 0.5 0.4
gén C 0.1 0.5 0 0.6 0.6
gén D 0.3 0.5 0.6 0 0.8
gén E 0.7 0.4 0.6 0.8 0
- Uvažujte motív reprezentovaný profilom (skórovaciou maticou, PSSM) uvedenou nižšie. Spočítajte skóre reťazca GGAG. Ktorá sekvencia dĺžky 4 bude mať najmenšie a ktorá najväčšie skóre?
A -3 3 -2 -2 C -2 -2 1 -2 G 0 -2 -1 3 T 1 -1 1 -2
- Nájdite všetky výskyty regulárneho výrazu TA[CG][AT]AT v sekvencii GACGATATAGTATGTACAATATGC.
Proteíny
Primárna, sekundárna a terciálna štruktúra proteínov, proteínové domény a rodiny, reprezentovanie rodiny pravdepodobnostným profilom a profilovým HMM, protein threading, gene ontology.
- Zostavte profil (PSSM) pre zarovnanie sekvencií uvedené nižšie, pričom predpokladáme, že v celej databáze A tvorí 60% a G 40% všetkých sekvencií (iné aminokyseliny neuvažujeme). Použite prirodzený logaritmus (ln) a pseudocount 1.
AAGA GAGA GAAA GGAG GGAA
RNA
Sekundárna štruktúra RNA, pseudouzol a dobre uzátvorkovaná štruktúra, Nussinovovej algoritmus, minimalizácia energie, stochastické bezkontextové gramatiky, kovariančné modely.
- Doplňte chýbajúce hodnoty za otázniky v matici dynamického programovania (Nussinovovej algoritmus) pre nájdenie najväčšieho počtu dobre uzátvorkovaných spárovaných báz v RNA sekvencii GAACUAUCUGA (dovoľujeme len komplementárne páry A-U, C-G) a nakreslite sekundárnu štruktúru, ktorú algoritmus našiel.
0 0 0 1 1 2 2 3 3 ? ?
0 0 0 1 1 2 2 3 3 ?
0 0 1 1 2 2 2 3 3
0 0 1 1 1 1 2 3
0 1 1 ? 1 2 3
0 1 1 1 2 2
0 0 0 1 2
0 0 1 1
0 0 1
0 0
0
- Uvažujme RNA sekvenciu dĺžky 27, ktorá má v sekundárnej štruktúre spárované komplementárne bázy na pozíciách: (2,23), (3,22), (4,21), (5,13), (6,12), (8,16), (9,15), (10,14), (18,26) a (19,25). Koľko najmenej párov z tohto zoznamu musíme odstrániť, aby sme dostali štruktúru bez pseudouzlov? Ktoré páry to budú?
- Uvažujme sekvenciu RNA ACUGAGUCCAAGG, ktorá má v sekundárnej štruktúre spárované bázy na pozíciách (1,7), (2,6), (3,5), (8,13) a (9,12). (Pozície číslujeme od 1.) (Táto RNA je uvedená ako príklad na strane 12 prednášky o RNA.) Ukážte akou postupnosťou pravidiel by sme ju mohli odvodiť v gramatike uvedenej nižšie tak, aby spárované bázy boli vždy vytvorené v jednom kroku odvodenia.
- Gramatika: S->aSu|uSa|cSg|gSc|aS|cS|gS|uS|Sa|Sc|Sg|Su|SS|epsilon
- Iný príklad gramatiky: S->aSu|uSa|cSg|gSc|TS|ST|SS|epsilon; T->aT|cT|gT|tT|epsilon
Populačná genetika
Polymorfizmus, SNP, alela, homozygot, heterozygot, rekombinácia, frekvencia polymorfizmu ako markovovský reťazec, náhodný genetický drift, väzbová nerovnováha (linkage disequilibrium), mapovanie asociácií, LD blok, subpopulácia.
- Pre dvojice SNPov, ktorých tabuľky sú uvedené nižšie, určite, či môžeme štatisticky vylúčiť hypotézu, že sú v stave väzbovej rovnováhy (LE, linkage equilibrium) pri hladine významnosti p=0.05, resp.
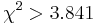 . Pre každú dvojicu spočítajte veličinu
. Pre každú dvojicu spočítajte veličinu 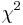 .
.
Q q Q q Q q
P 100 200 P 10 20 P 1 2
p 300 200 p 30 20 p 3 2
Ďalšie dôležité znalosti z cvičení pre informatikov
(iba informatická časť skúšky)
- Pokročilejšie ukážky dynamického programovania (proteíny MS/MS, varianty zarovnávania sekvencií, varianty Nussinovovej algoritmu)
- BLAST, MinHashing
- Algoritmy pre použitie HMM (Viterbiho, dopredný)
- Felsensteinov algoritmus
- Celočíselné lineárne programovanie
- EM algoritmus na hľadanie motívov
Ďalšie dôležité znalosti z cvičení pre biológov
(iba biologická časť skúšky)
- Interpretácia dotplotov
- Interpretácia fylogenetických stromov, bootstrap, zakorenenie
- Interpretácia vizualizácií z UCSC genome browsera
- Ukážky rôznych bioinformatických programov, súvis ich nastavení a výsledkov s pojmami z prednášky
- Analýza nadreprezentácie, multiple testing correction, K-means clustering