CB04: Rozdiel medzi revíziami
(Vytvorená stránka „==Praktické cvičenie pri počítači: dotploty== ===Yass a dotploty=== * Program Yass hlada lokalne zarovnania v DNA sekvenciach, zobrazuje vo forme dot plotov * V nov...“) |
|||
| Riadok 1: | Riadok 1: | ||
| + | ===Pravdepodobnostný model náhodnej sekvencie=== | ||
| + | * Napríklad chceme modelovať náhodnú DNA sekvenciu dĺžky n s obsahom GC 40% | ||
| + | * Máme vrece s guľôčkami označenými A,C,G,T, pričom guľôčok označených A je 30%, C 20%, G 20% a T 30%. | ||
| + | * Vytiahneme guľôčku, zapíšeme si písmeno, hodíme ju naspäť, zamiešame a opakujeme s ďalším písmenom atď, až kým nevygenerujeme n písmen | ||
| + | |||
| + | * Vytiahnime z mechu 2x guľôčku. Prvé písmeno, ktoré nám vyjde, označme X1 a druhé X2 | ||
| + | * Pr(X1=A) = 0.3, Pr(X2=C)=0.2 | ||
| + | * Pr(X1=A a X2=C) = Pr(X1=A)*Pr(X2=C) = 0.3*0.2 = 0.06 | ||
| + | ** T.j. šanca, že dostaneme sekvenciu AC po dvoch ťahoch je 6% | ||
| + | ** Ak rátame pravdepodobnosť, že sa dve nezávislé udalosti stanú súčasne, ich pravdepodobnosti násobíme. V tomto prípade to, či X1=A je nezávislé od toho, či X2=C | ||
| + | * Pr(X1 je A alebo C) = Pr(X1=A)+Pr(X1=C) = 0.3+0.2 = 0.5 | ||
| + | ** Pravdepodobnosť, že prvé písmeno bude A alebo C je 50% | ||
| + | ** Pravdepodobnosti navzájom sa vylučujúcich udalostí (X1=A a X1=C) sa môžu sčítať, čím dostaneme pravdepodobnosť, že aspoň jedna z nich nastane | ||
| + | * Pr(v sekvencii je aspoň jedno A) = Pr(X1=A alebo X2=A) nemôžeme počítať ako Pr(X1=A)+Pr(X2=A), lebo sa navzájom nevylučujú a prípad, že X1=A a X2=A by sme započítali dvakrát | ||
| + | * Správne je Pr(X1 je A alebo X2 je A) = Pr(X1=A) + Pr(X1 <> A a X2=A) = Pr(X1=A) + Pr(X1 <> A) * Pr(X2=A) = 0.3+0.7*0.3 = 0.51 | ||
| + | * Pr(X1=X2) = Pr(X1=X2=A) + Pr(X1=X2=C) + Pr(X1=X2=G) + Pr(X1=X2=T) = 0.3*0.3+0.2*0.2+0.2*0.2+0.3*0.3 = 0.26. | ||
| + | * Ak u označíme pravdepodobnosť u = Pr(X1=A)=Pr(X1=T)=Pr(X2=A)=Pr(X2=T) a v=Pr(X1=C)=Pr(X1=G)=Pr(X2=C)=Pr(X2=G), aký bude vzorec pre Pr(X1=X2)? | ||
| + | |||
| + | ==Skórovacie matice== | ||
| + | |||
| + | Chceme určiť skórovaciu schému pre zarovnávanie dvoch DNA sekvencií (bez medzier). Máme dva modely, každý z nich vie vygenerovať 2 zarovnané sekvencie dĺžky ''n''. | ||
| + | |||
| + | '''Model R (random)''' reprezentuje nezávislé náhodne sekvencie | ||
| + | * Použijeme naše vrece s guličkami označenými A,C,G,T, pričom guličiek označených A je 30%, C 20%, G 20% a T 30%. | ||
| + | * Vytiahneme guličku, zapíšeme si písmeno, hodíme ju naspäť, zamiešame a opakujeme s ďalším písmenom atď až kým nevygenerujeme ''n'' písmen pre jednu sekvenciu a ''n'' písmen pre druhú | ||
| + | * Máme jednu sekvenciu ACT a druhú ACC. Aká je pravdepodobnosť, že práve tieto sekvencie vygenerujeme v našom modeli R? | ||
| + | * Nezávislé udalosti pre jednotlivé písmená, t.j. Pr(X1=A)*Pr(X2=C)*Pr(X3=T)*Pr(Y1=A)*Pr(Y2=C)*Pr(Y3=C) = 0.3*0.2*0.3*0.3*0.2*0.2 = 0.000216 | ||
| + | * Spolu máme v modeli <math>4^6 = 4096</math> možností ako vygenerovať 2 DNA sekvencie dĺžky 3 | ||
| + | |||
| + | '''Model H (homolog)''' reprezentuje zarovnanie vzájomne súvisiacich sekvencií | ||
| + | * máme vrece, v ktorom je napr. | ||
| + | ** po 21% guličiek označených AA, TT | ||
| + | ** po 14% označených CC, GG | ||
| + | ** po 2.4% označených AC, AG, CA, CT, GA, GT, TC, TG | ||
| + | ** po 3.6% označených AT, TA | ||
| + | ** po 1.6% označených CG, GC. | ||
| + | * Spolu máme 70% guličiek označených rovnakými písmenami, 30% rôznymi | ||
| + | |||
| + | * ''n'' krát z vreca vytiahneme guličku a písmená píšeme ako stĺpce zarovnania A1, A2,.., An. | ||
| + | * aká je pravdepodobnosť, ze dostaneme ACT zarovnané s ACC? | ||
| + | * Pr(A1=AA)*Pr(A2=CC)*Pr(A3=TC) = 0.21*0.14*0.024 = 0.0007056 | ||
| + | |||
| + | '''Skóre zarovnania''' je log Pr(zarovnania v H)/Pr(zarovnania v R), t.j. log (0.0007056 / 0.000216) = 0.514105 (pre desiatkový logaritmus) | ||
| + | * kladné skóre znamená, že model H lepšie zodpovedá dátam (zarovnaniu) ako model R | ||
| + | * záporné skóre znamená, že model R lepšie zodpovedá dátam | ||
| + | |||
| + | ===Cvičenie pri počítači=== | ||
| + | * Stiahnite si súbor [http://compbio.fmph.uniba.sk/vyuka/mbi-data/cb05/scoring.ods], uložte si ho a otvorte v Openoffice | ||
| + | * V záložke Matica vyplňte do žltej oblasti vzorce na výpočet pravdepodobnosti krátkeho zarovnania, logaritmus pomeru pravdepodobnosti a súčet skóre, pričom vo vzorcoch použijete odkazy na políčka v riadkoch 9-13, stĺpcoch B a E | ||
| + | * Súčet skóre by mal zhruba rovný desaťnásobku logaritmu pomeru - prečo vidíme rozdiely? | ||
| + | |||
| + | * Potom skúšajte meniť %GC a %identity v horných riadkoch tabuľky a pozrite sa, ako to ovplyvní skórovanie. Výsledné skóre zo stĺpca E ručne prepíšte (bez formúl) do tabuľky v záložke Výsledky. Prečo nastávajú také zmeny ako vidíte? | ||
| + | |||
| + | |||
==Praktické cvičenie pri počítači: dotploty== | ==Praktické cvičenie pri počítači: dotploty== | ||
===Yass a dotploty=== | ===Yass a dotploty=== | ||
| Riadok 192: | Riadok 246: | ||
CCGAAAAGCCCCCACAAAAAGCCG | CCGAAAAGCCCCCACAAAAAGCCG | ||
</pre> | </pre> | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
Verzia zo dňa a času 14:14, 15. október 2020
Obsah
Pravdepodobnostný model náhodnej sekvencie
- Napríklad chceme modelovať náhodnú DNA sekvenciu dĺžky n s obsahom GC 40%
- Máme vrece s guľôčkami označenými A,C,G,T, pričom guľôčok označených A je 30%, C 20%, G 20% a T 30%.
- Vytiahneme guľôčku, zapíšeme si písmeno, hodíme ju naspäť, zamiešame a opakujeme s ďalším písmenom atď, až kým nevygenerujeme n písmen
- Vytiahnime z mechu 2x guľôčku. Prvé písmeno, ktoré nám vyjde, označme X1 a druhé X2
- Pr(X1=A) = 0.3, Pr(X2=C)=0.2
- Pr(X1=A a X2=C) = Pr(X1=A)*Pr(X2=C) = 0.3*0.2 = 0.06
- T.j. šanca, že dostaneme sekvenciu AC po dvoch ťahoch je 6%
- Ak rátame pravdepodobnosť, že sa dve nezávislé udalosti stanú súčasne, ich pravdepodobnosti násobíme. V tomto prípade to, či X1=A je nezávislé od toho, či X2=C
- Pr(X1 je A alebo C) = Pr(X1=A)+Pr(X1=C) = 0.3+0.2 = 0.5
- Pravdepodobnosť, že prvé písmeno bude A alebo C je 50%
- Pravdepodobnosti navzájom sa vylučujúcich udalostí (X1=A a X1=C) sa môžu sčítať, čím dostaneme pravdepodobnosť, že aspoň jedna z nich nastane
- Pr(v sekvencii je aspoň jedno A) = Pr(X1=A alebo X2=A) nemôžeme počítať ako Pr(X1=A)+Pr(X2=A), lebo sa navzájom nevylučujú a prípad, že X1=A a X2=A by sme započítali dvakrát
- Správne je Pr(X1 je A alebo X2 je A) = Pr(X1=A) + Pr(X1 <> A a X2=A) = Pr(X1=A) + Pr(X1 <> A) * Pr(X2=A) = 0.3+0.7*0.3 = 0.51
- Pr(X1=X2) = Pr(X1=X2=A) + Pr(X1=X2=C) + Pr(X1=X2=G) + Pr(X1=X2=T) = 0.3*0.3+0.2*0.2+0.2*0.2+0.3*0.3 = 0.26.
- Ak u označíme pravdepodobnosť u = Pr(X1=A)=Pr(X1=T)=Pr(X2=A)=Pr(X2=T) a v=Pr(X1=C)=Pr(X1=G)=Pr(X2=C)=Pr(X2=G), aký bude vzorec pre Pr(X1=X2)?
Skórovacie matice
Chceme určiť skórovaciu schému pre zarovnávanie dvoch DNA sekvencií (bez medzier). Máme dva modely, každý z nich vie vygenerovať 2 zarovnané sekvencie dĺžky n.
Model R (random) reprezentuje nezávislé náhodne sekvencie
- Použijeme naše vrece s guličkami označenými A,C,G,T, pričom guličiek označených A je 30%, C 20%, G 20% a T 30%.
- Vytiahneme guličku, zapíšeme si písmeno, hodíme ju naspäť, zamiešame a opakujeme s ďalším písmenom atď až kým nevygenerujeme n písmen pre jednu sekvenciu a n písmen pre druhú
- Máme jednu sekvenciu ACT a druhú ACC. Aká je pravdepodobnosť, že práve tieto sekvencie vygenerujeme v našom modeli R?
- Nezávislé udalosti pre jednotlivé písmená, t.j. Pr(X1=A)*Pr(X2=C)*Pr(X3=T)*Pr(Y1=A)*Pr(Y2=C)*Pr(Y3=C) = 0.3*0.2*0.3*0.3*0.2*0.2 = 0.000216
- Spolu máme v modeli
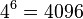 možností ako vygenerovať 2 DNA sekvencie dĺžky 3
možností ako vygenerovať 2 DNA sekvencie dĺžky 3
Model H (homolog) reprezentuje zarovnanie vzájomne súvisiacich sekvencií
- máme vrece, v ktorom je napr.
- po 21% guličiek označených AA, TT
- po 14% označených CC, GG
- po 2.4% označených AC, AG, CA, CT, GA, GT, TC, TG
- po 3.6% označených AT, TA
- po 1.6% označených CG, GC.
- Spolu máme 70% guličiek označených rovnakými písmenami, 30% rôznymi
- n krát z vreca vytiahneme guličku a písmená píšeme ako stĺpce zarovnania A1, A2,.., An.
- aká je pravdepodobnosť, ze dostaneme ACT zarovnané s ACC?
- Pr(A1=AA)*Pr(A2=CC)*Pr(A3=TC) = 0.21*0.14*0.024 = 0.0007056
Skóre zarovnania je log Pr(zarovnania v H)/Pr(zarovnania v R), t.j. log (0.0007056 / 0.000216) = 0.514105 (pre desiatkový logaritmus)
- kladné skóre znamená, že model H lepšie zodpovedá dátam (zarovnaniu) ako model R
- záporné skóre znamená, že model R lepšie zodpovedá dátam
Cvičenie pri počítači
- Stiahnite si súbor [1], uložte si ho a otvorte v Openoffice
- V záložke Matica vyplňte do žltej oblasti vzorce na výpočet pravdepodobnosti krátkeho zarovnania, logaritmus pomeru pravdepodobnosti a súčet skóre, pričom vo vzorcoch použijete odkazy na políčka v riadkoch 9-13, stĺpcoch B a E
- Súčet skóre by mal zhruba rovný desaťnásobku logaritmu pomeru - prečo vidíme rozdiely?
- Potom skúšajte meniť %GC a %identity v horných riadkoch tabuľky a pozrite sa, ako to ovplyvní skórovanie. Výsledné skóre zo stĺpca E ručne prepíšte (bez formúl) do tabuľky v záložke Výsledky. Prečo nastávajú také zmeny ako vidíte?
Praktické cvičenie pri počítači: dotploty
Yass a dotploty
- Program Yass hlada lokalne zarovnania v DNA sekvenciach, zobrazuje vo forme dot plotov
- V novom okne/tabe si otvorte YASS server na adrese http://bioinfo.lifl.fr/yass/yass.php
- V dalsom okne si na stranke UCSC genome browseru si zobrazte oblast chr21:9,180,027-9,180,345 vo verzii hg38 ludskeho genomu [2]
- tento región obsahuje Alu repeat. Tieto opakovania tvoria cca 10% ľudského genómu, viac ako milión kópií
- zobrazte si DNA sekvenciu tohto useku takto: na hornej modrej liste zvolte View, potom v podmenu DNA, na dalsej obrazovke tlacidlo get DNA
- DNA sekvenciu Alu opakovania chceme zarovnat samu k sebe programom YASS
- DNA sekvenciu Alu opakovania skopirujte do okienka "Paste your sequences" v stranke Yass-u a dvakrat stlacte tlacidlo Select vedla okienka
- Nizsie v casti "Selected DNA sequence(s)" by sa Vam malo v oboch riadkoch objavit "Pasted file 1"
- Nizsie v casti "Parameters" zvolte "E-value threshold" 0.01 a stlacte "Run YASS"
- Vo vysledkoch si pozrite Dotplot, co z neho viete usudit o podobnosti jednotlivych casti Alu opakovania?
- Vo vysledkoch si pozrite Raw: blast, ake su suradnice opakujucej sa casti a kolko zarovnanie obsahuje zhod/nezhod/medzier? (Pozor, prve zarovnanie je cela sekvencia sama k sebe, druhe je asi to, co chcete)
- V genome browseri sa presunte na poziciu chr21:8,552,000-8,562,000 (10kb sekvencie na chromozome 21, s niekolkymi vyskytmi Alu) [3]
- Chceme teraz porovnat tento usek genomu so sekvenciou Alu pomocou YASSu
- Ako predtym si stiahnite DNA sekvenciu tohto useku
- V YASSe chodte sipkou spat na formular
- Skopirujte DNA sekvenciu do YASSoveho formulara, do okienka vpravo (vyznacit si ju mozete klavesovou kombinaciou Ctrl-A alebo Select All v menu Edit),
- V casti formulara Selected DNA sequence(s) stlacte Remove pri hornom riadku
- Pri pravom okienku, kam ste nakopirovali sekvenciu, stlacte Select
- Zase stlacte Run YASS
- Pozrite si vysledok ako Dotplot, kolko opakovani Alu ste nasli? Preco je jedno cervene?
- Pozrite si Raw: blast, na kolko percent sa podoba najpodobnejsia a na kolko druha najpodobnejsia kopia?
Príklady praktických programov
Pozrime sa na niekolko nastrojov, vsimnime si, ake poskytuju nastavenia a co vypisuju na vystupe, dajme to do suvisu s prednaskami
- viacnasobne zarovnania neskor
Plné dynamické programovanie
- Balicek emboss, obsahuje programy na klasicke dynamicke programovanie (needle - globalne, water - lokalne), najdu sa na stranke EBI http://www.ebi.ac.uk/Tools/psa/
- porovnanie lokalneho a globalneho zarovnania
- Dva proteiny s kinase doménou zarovnáme lokálne, globálne a globálne s tým, že neplatíme za medzery na koncoch
>sp|P50520|VPS34_SCHPO Phosphatidylinositol 3-kinase vps34 OS=Schizosaccharomyces pombe (strain 972 / ATCC 24843) GN=vps34 PE=2 SV=2 >tr|B1AKP8|B1AKP8_HUMAN FK506 binding protein 12-rapamycin associated protein 1 OS=Homo sapiens GN=FRAP1 PE=4 SV=1
- sekvencie a vysledne zarovnania: #CB-aln-dp
- vo vysledku si vsimnime, kolko ma kazde z nich %identity, %gaps, a kam sa zarovna sekvencia na pozicii 53 v spodnej sekvencii (NSESEAE) a kam sekvencia na pozicii 395 (EDLRQDE)
Lokalne zarovnanie
Length: 645
Identity: 124/645 (19.2%)
Similarity: 221/645 (34.3%)
Gaps: 211/645 (32.7%)
Score: 226.0
VPS34_SCHPO 235-738
B1AKP8_HUMAN 53-627
235 NLDSPAE
|.:|.||
53 NSESEAE
549 DDLRQDQ
:|||||:
395 EDLRQDE
Globalne zarovnanie
Length: 948
Identity: 167/948 (17.6%)
Similarity: 292/948 (30.8%)
Gaps: 341/948 (36.0%)
Score: 130.5
VPS34_SCHPO 1-801
B1AKP8_HUMAN 1-754
102 NDEEVYE
|.|...|
53 NSESEAE
549 DDLRQDQ
:|||||:
395 EDLRQDE
Globalne zarovnanie s nulovou penaltou za medzeru na koncoch
Length: 1060
Identity: 138/1060 (13.0%)
Similarity: 245/1060 (23.1%)
Gaps: 565/1060 (53.3%)
Score: 206.0
VPS34_SCHPO 234-801
B1AKP8_HUMAN 1-674
265 KIRKELESIL
....|.||..
53 NSESEAESTE
549 DDLRQDQ
:|||||:
395 EDLRQDE
NCBI Blast
- NCBI BLAST http://blast.ncbi.nlm.nih.gov/ vela roznych nastrojov (porovnavanie DNA vs proteiny, pripadne translacia DNA na protein v 6 ramcoch)
- Heuristicky algoritmus, moze niektore zarovnania vynechat
- rozne nastavenia, vystup E-value
Low complexity masking: nepouzivat pri hladani jadier zarovnania regiony v ktorych sa velakrat opakuje ta ista aminokyselina
- Priklad (z ucebnice Zvelebil and Baum):
>sp|P04156|PRIO_HUMAN Major prion protein OS=Homo sapiens GN=PRNP PE=1 SV=1 MANLGCWMLVLFVATWSDLGLCKKRPKPGGWNTGGSRYPGQGSPGGNRYPPQGGGGWGQP HGGGWGQPHGGGWGQPHGGGWGQPHGGGWGQGGGTHSQWNKPSKPKTNMKHMAGAAAAGA VVGGLGGYMLGSAMSRPIIHFGSDYEDRYYRENMHRYPNQVYYRPMDEYSNQNNFVHDCV NITIKQHTVTTTTKGENFTETDVKMMERVVEQMCITQYERESQAYYQRGSSMVLFSSPPV ILLISFLIFLIVG
- Hladajme v databaze Reference sequence (Refseq), organizmus human
Bez maskovania vypise napr aj toto zarovnanie:
>ref|NP_065842.1| serine/threonine-protein kinase TAO1 isoform 1 [Homo sapiens]
Length=1001
Score = 45.1 bits (105), Expect = 1e-06, Method: Composition-based stats.
Identities = 26/61 (43%), Positives = 27/61 (44%), Gaps = 11/61 (18%)
Query 38 YPGQGSPGGNRYPPQGGGG--WGQPHGG---GWGQPHGGG---WGQPHGGGWGQPHGGGWG 90
YPG G + P GG G WG P GG WG P GG WG P G G P G G
Sbjct 904 YPGAS---GWSHNPTGGPGPHWGHPMGGPPQAWGHPMQGGPQPWGHPSGPMQGVPRGSSMG 961
Score = 40.0 bits (92), Expect = 4e-05, Method: Composition-based stats.
Identities = 25/62 (40%), Positives = 25/62 (40%), Gaps = 10/62 (16%)
Query 26 PKPGGW--NTGGSRYPGQGSPGGNRYPPQGGGGWGQPHGGG---WGQPHGGGWGQPHGGGWG 82
P GW N G P G P G PPQ WG P GG WG P G G P G
Sbjct 905 PGASGWSHNPTGGPGPHWGHPMGG--PPQA---WGHPMQGGPQPWGHPSGPMQGVPRGSSMG 961
Ak zapneme maskovanie, toto zarovnanie uz nenajde, v zarovnani sameho so sebou sa objavia male pismena alebo Xka:
>ref|NP_000302.1|major prion protein preproprotein [Homo sapiens]
Length=253
Score = 520 bits (1340), Expect = 0.0, Method: Compositional matrix adjust.
Identities = 253/253 (100%), Positives = 253/253 (100%), Gaps = 0/253 (0%)
Query 1 MANLGCWMLVLFVATWSDLGLCKKRPKPGGWNTGGSRYPGQGSPGGNRYppqggggwgqp 60
MANLGCWMLVLFVATWSDLGLCKKRPKPGGWNTGGSRYPGQGSPGGNRYPPQGGGGWGQP
Sbjct 1 MANLGCWMLVLFVATWSDLGLCKKRPKPGGWNTGGSRYPGQGSPGGNRYPPQGGGGWGQP 60
Query 61 hgggwgqphgggwgqphgggwgqphgggwgqgggTHSQWNKPSKPKTNMKHMagaaaaga 120
HGGGWGQPHGGGWGQPHGGGWGQPHGGGWGQGGGTHSQWNKPSKPKTNMKHMAGAAAAGA
Sbjct 61 HGGGWGQPHGGGWGQPHGGGWGQPHGGGWGQGGGTHSQWNKPSKPKTNMKHMAGAAAAGA 120
Query 121 vvgglggymlgsamsRPIIHFGSDYEDRYYRENMHRYPNQVYYRPMDEYSNQNNFVHDCV 180
VVGGLGGYMLGSAMSRPIIHFGSDYEDRYYRENMHRYPNQVYYRPMDEYSNQNNFVHDCV
Sbjct 121 VVGGLGGYMLGSAMSRPIIHFGSDYEDRYYRENMHRYPNQVYYRPMDEYSNQNNFVHDCV 180
Query 181 NITIKQHtvttttkgenftetDVKMMERVVEQMCITQYERESQAYYQRGSSMVLFSsppv 240
NITIKQHTVTTTTKGENFTETDVKMMERVVEQMCITQYERESQAYYQRGSSMVLFSSPPV
Sbjct 181 NITIKQHTVTTTTKGENFTETDVKMMERVVEQMCITQYERESQAYYQRGSSMVLFSSPPV 240
Query 241 illisfliflivG 253
ILLISFLIFLIVG
Sbjct 241 ILLISFLIFLIVG 253
BLAT, chains, nets v UCSC browseri
- Program BLAT v UCSC browseri http://genome-euro.ucsc.edu/ rychlo vyhladava sekvencie v genome, ale nevie najst slabsie podobnosti
- Vhodne pouzitie: zarovnanie mRNA ku genomu, presne urcenie suradnic nejakej sekvencie, a pod.
- Net tracky v UCSC genome browseri nam umoznuju prechadzat medzi homologickymi oblastami roznych genomov
Praktické cvičenie pri počítači: BLAT vs BLAST
BLAT/BLAST
- Sekvencia uvedena nizsie vznikla pomocou RT-PCR na ľudských cDNA knižniciach
- Choďte na UCSC genome browser http://genome-euro.ucsc.edu/ , na modrej lište zvoľte BLAT, zadajte túto sekvenciu a hľadajte ju v ľudskom genóme. Akú podobnosť (IDENTITY) má najsilnejší nájdený výskyt? Aký dlhý úsek genómu zasahuje? (SPAN). Všimnite si, že ostatné výskyty sú oveľa kratšie.
- V stĺpci ACTIONS si pomocou Details môžete pozrieť detaily zarovnania a pomocou Browser si pozrieť príslušný úsek genómu.
- V tomto úseku genómu si zapnite track Vertebrate net na full a kliknutím na farebnú čiaru na obrázku pre tento track zistite, na ktorom chromozóme sliepky sa vyskytuje homologický úsek.
- Skusme tu istu sekvenciu zarovnat ku genomu sliepky programom Blat: stlacte najprv na hornej modrej liste Genomes, zvolte Vertebrates a Chicken a potom na hornej liste BLAT. Do okienka zadajte tu istu sekvenciu. Akú podobnosť a dĺžku má najsilnejší nájdený výskyt teraz? Na ktorom je chromozóme?
- Skúsme to isté v NCBI blaste: Choďte na http://blast.ncbi.nlm.nih.gov/ zvoľte nucleotide blast, database others a z menu reference genomic sequence, organism chicken (taxid:9031), program blastn
- Aká je dĺžka, identity a E-value najlepšieho zarovnania? Na ktorom je chromozóme?
RT PCR sekvencia z cvičenia vyššie
AACCATGGGTATATACGACTCACTATAGGGGGATATCAGCTGGGATGGCAAATAATGATTTTATTTTGAC TGATAGTGACCTGTTCGTTGCAACAAATTGATAAGCAATGCTTTCTTATAATGCCAACTTTGTACAAGAA AGTTGGGCAGGTGTGTTTTTTGTCCTTCAGGTAGCCGAAGAGCATCTCCAGGCCCCCCTCCACCAGCTCC GGCAGAGGCTTGGATAAAGGGTTGTGGGAAATGTGGAGCCCTTTGTCCATGGGATTCCAGGCGATCCTCA CCAGTCTACACAGCAGGTGGAGTTCGCTCGGGAGGGTCTGGATGTCATTGTTGTTGAGGTTCAGCAGCTC CAGGCTGGTGACCAGGCAAAGCGACCTCGGGAAGGAGTGGATGTTGTTGCCCTCTGCGATGAAGATCTGC AGGCTGGCCAGGTGCTGGATGCTCTCAGCGATGTTTTCCAGGCGATTCGAGCCCACGTGCAAGAAAATCA GTTCCTTCAGGGAGAACACACACATGGGGATGTGCGCGAAGAAGTTGTTGCTGAGGTTTAGCTTCCTCAG TCTAGAGAGGTCGGCGAAGCATGCAGGGAGCTGGGACAGGCAGTTGTGCGACAAGCTCAGGACCTCCAGC TTTCGGCACAAGCTCAGCTCGGCCGGCACCTCTGTCAGGCAGTTCATGTTGACAAACAGGACCTTGAGGC ACTGTAGGAGGCTCACTTCTCTGGGCAGGCTCTTCAGGCGGTTCCCGCACAAGTTCAGGACCACGATCCG GGTCAGTTTCCCCACCTCGGGGAGGGAGAACCCCGGAGCTGGTTGTGAGACAAATTGAGTTTCTGGACCC CCGAAAAGCCCCCACAAAAAGCCG