CI10
Z MBI
Obsah
Úvod do bioinformatických databáz a on-line nástrojov
NCBI, Genbank, Pubmed, blast
- National Center for Biotechnology Information http://www.ncbi.nlm.nih.gov/
- Zhromazduje verejne pristupne data z molekularnej biologie
- Mozeme hladat klucove slova v roznych databazach
- Pubmed: databaza clankov, napr. najdime phastcons
- Gene: najdime DNA polymerase
- BLAST: najdime nasledujucu sekvenciu v genome kurata (zvoľme nucleotide blast, database others a z menu reference genomic sequence, organism chicken (taxid:9031), program blastn)
- Ide o osekvenovany kusok ludskej mRNA, kde v kuracom genome sme nasli homolog, ake ma dlzku, skore, E-value, % zhodnych baz?
AACCATGGGTATATACGACTCACTATAGGGGGATATCAGCTGGGATGGCAAATAATGATTTTATTTTGAC TGATAGTGACCTGTTCGTTGCAACAAATTGATAAGCAATGCTTTCTTATAATGCCAACTTTGTACAAGAA AGTTGGGCAGGTGTGTTTTTTGTCCTTCAGGTAGCCGAAGAGCATCTCCAGGCCCCCCTCCACCAGCTCC GGCAGAGGCTTGGATAAAGGGTTGTGGGAAATGTGGAGCCCTTTGTCCATGGGATTCCAGGCGATCCTCA CCAGTCTACACAGCAGGTGGAGTTCGCTCGGGAGGGTCTGGATGTCATTGTTGTTGAGGTTCAGCAGCTC CAGGCTGGTGACCAGGCAAAGCGACCTCGGGAAGGAGTGGATGTTGTTGCCCTCTGCGATGAAGATCTGC AGGCTGGCCAGGTGCTGGATGCTCTCAGCGATGTTTTCCAGGCGATTCGAGCCCACGTGCAAGAAAATCA GTTCCTTCAGGGAGAACACACACATGGGGATGTGCGCGAAGAAGTTGTTGCTGAGGTTTAGCTTCCTCAG TCTAGAGAGGTCGGCGAAGCATGCAGGGAGCTGGGACAGGCAGTTGTGCGACAAGCTCAGGACCTCCAGC TTTCGGCACAAGCTCAGCTCGGCCGGCACCTCTGTCAGGCAGTTCATGTTGACAAACAGGACCTTGAGGC ACTGTAGGAGGCTCACTTCTCTGGGCAGGCTCTTCAGGCGGTTCCCGCACAAGTTCAGGACCACGATCCG GGTCAGTTTCCCCACCTCGGGGAGGGAGAACCCCGGAGCTGGTTGTGAGACAAATTGAGTTTCTGGACCC CCGAAAAGCCCCCACAAAAAGCCG
Uniprot
- Prehladnejsi pohlad na proteiny, vela linkov na ine databazy, cast vytvarana rucne
- Pozrieme sa na známy koronavírusový proteín Spike
- Nájdime ho na stránke http://www.uniprot.org/ pod názvom SPIKE_SARS2
- Pozrime si podrobne jeho stránku, ktoré časti boli predpovedané bioinformatickými metódami z prednášky?
- Všimnime si niektorú Pfam doménu a pozrime si jej stránku
UCSC genome browser
- http://genome-euro.ucsc.edu/
- On-line grafický nástroj na prezeranie genómov
- Konfigurovateľný, veľa možností, prijemne pouzivatelske rozhranie
- Moznost stiahnut data vhodne na dalsie spracovanie alebo zobrazit vlastne data
- Pomerne málo organizmov
- doraz hlavne na ludsky genom
Základy
- Adresa http://genome.ucsc.edu/
- Hore v modrom menu zvoľte Genomes, potom zvoľte ľudský genóm. Do okienka search term zadajte HOXA2. Vo výsledkoch hľadania (UCSC genes) zvoľte gén homeobox A2 na chromozóme 7.
- Pozrime si spolu túto stránku
- V hornej časti sú ovládacie prvky na pohyb vľavo, vpravo, približovanie, vzďaľovanie
- Pod tým schéma chromozómu, červeným vyznačená zobrazená oblasť
- Pod tým obrázok vybranej oblasti, rôzne tracky
- Pod tým zoznam všetkých trackov, dajú sa zapínať, vypínať a konfigurovať
- Po kliknutí na obrázok sa často zobrazí ďalšia informácia o danom géne alebo inom zdroji dát
- V génoch exony hrubé, UTR tenšie, intróny vodorovné čiary
- Po kliknutí na gén alebo inú časť nejakého tracku väčšinou o ňom dostaneme viac informácií. Kliknutim na listu ku tracku (lavy okraj obazku) sa dozviete viac o tracku a mozete nastavovat parametre zobrazenia
Sekvenovanie
- Hore v modrom menu zvoľte Genomes
- Na ďalšej stránke zvoľte človeka a v menu Assembly zistite, kedy boli pridané posledné dve verzie ľudského genómu (hg19 a hg38)
- Na tej istej stránke dole nájdete stručný popis zvolenej verzie genómu. Pre ktoré oblasti genómu máme v hg38 najviac alternatívnych verzií?
- Zadajte región chr21:31,250,000-31,300,000 v hg19 [1]
- Zapnite si tracky Mapability a RepeatMasker na "full"
- Mapability: nakoľko sa daný úsek opakuje v genóme a či teda vieme jednoznačne jeho ready namapovať pri použití Next generation sequencing
- Ako a prečo sa pri rôznych dĺžkach readov líšia? (Keď kliknete na linku "Mapability", môžete si prečítať bližšie detaily.)
- Približne v strede zobrazeného regiónu je pokles mapovateľnosti. Akému typu opakovania zodpovedá? (pozrite track RepeatMasker)
- Zapnite si tracky "Assembly" a "Gaps" a pozrite si región chr2:110,000,000-110,300,000 v hg19. [2] Aká dlhá je neosekvenovaná medzera (gap) v strede tohto regiónu? Približnú veľkosť môžete odčítať z obrázku, presnejší údaj zistíte kliknutím na čierny obdĺžnik zodpovedajúci tejto medzere (úplne presná dĺžka aj tak nebola známa, nakoľko nebola osekvenovaná).
- Cez menu položku View, In other genomes si pozrite, ako zobrazený úsek vyzerá vo verzii hg38. Ako sa zmenila dĺžka z pôvodných 300kb?
- Prejdite na genóm Rhesus, verzia rheMac2, región chr7:59,022,000-59,024,000 [3], zapnite si tracky Contigs, Gaps, Quality scores
- Aké typy problémov v kvalite sekvencie v tomto regióne vidíte?
Komparativna genomika
- V casti multiz alignments vidite zarovnania k roznym inym genomom (da sa zapinat, ze ku ktorym). Mozete si pozriet, ako sa uroven zarovnania zmeni ked sa priblizujeme a vzdalujeme (zoom in/zoom out).
- Ked sa priblizite na uroven "base", t.j. zobrazenych cca 100bp, v obdlzniku multiz alignment uvidite zarovnanie s homologickym usekom v inych genomoch.
- V casti conservation by PhyloP vidime graf toho, ako silne su zachovane jednotlive stlpce zarovnania
- Da sa zapnut track Placental Chain/Net a pozriet sa na ktorych chromozomoch je ortologicky usek v inych genomoch
Blat
- Choďte na UCSC genome browser (http//genome.ucsc.edu/), na modrej lište zvoľte BLAT, zadajte DNA sekvenciu vyssie a hľadajte ju v ľudskom genóme. Akú podobnosť (IDENTITY) má najsilnejší nájdený výskyt? Aký dlhý úsek genómu zasahuje? (SPAN). Všimnite si, že ostatné výskyty sú oveľa kratšie.
- V stĺpci ACTIONS si pomocou Details môžete pozrieť detaily zarovnania a pomocou Browser si pozrieť príslušný úsek genómu.
- V tomto úseku genómu si zapnite track Vertebrate net na full a kliknutím na farebnú čiaru na obrázku pre tento track zistite, na ktorom chromozóme kuraťa sa vyskytuje homologický úsek.
- Skusme tu istu sekvenciu namapovat do genomu sliepky: stlacte najprv na hornej modrej liste Genomes, zvolte Vertebrates a Chicken a potom na hornej liste BLAT. Do okienka zadajte tu istu sekvenciu. Akú podobnosť a dĺžku má najsilnejší nájdený výskyt teraz? Na ktorom je chromozóme?
- Ako sa to porovna s hodnotami, ktore sme dostali pomocou BLASTu na NCBI?
Práca s tabuľkami, sťahovanie anotácií
- Položka Tables na hornej lište umožnuje robiť rafinované veci s tabuľkami, ktoré obsahujú súradnice génov a pod.
- Základná vec: vyexportovať napr. všetky gény v zobrazenom výseku v niektorom formáte:
- sequence: fasta súbor proteínov, génov alebo mRNA s rôznymi nastaveniami
- GTF: súradnice
- Hyperlinks to genome browser: klikacia stránka
- Namiesto exportu si môžeme pozrieť rôzne štatistiky
- Zložitejšie: prienik dvoch tabuliek, napr. gény, ktoré sú viac než 50% pokryté simple repeats
- V intersection zvolíme group: Variation and repeats, track: RepeatMasker, nastavíme records that have at least 50% overlap with RepeatMasker
- V summary/statistics zistíme, kolko ich je v genóme, môžeme si ich preklikať cez Hyperlinks to genome browser
- Filter na tabuľku, napr. gény, ktoré majú v názve ribosomal (postup pre drozofilu):
- V casti hg19.kgXref based filters políčko description dáme *ribosomal*
Úvod do bezkontextových gramatík
- Na modelovanie štruktúry RNA sa používajú stochastické bezkontextové gramatiky (bude na ďalšej prednáške)
- Tie sú založené na bezkontextových gramatikách, ktoré mnohí poznáte z bakalárskeho štúdia
Gramatika
- Príklad: S->aSb, S->epsilon (píšeme aj skrátene S->aSb|epsilon)
- Dva typy symbolov: terminály (malé písmená), neterminály (veľké písmená)
- Pravidlá prepisujúce neterminál na reťazec terminálov a neterminálov (môže byť aj prázdny reťazec, ktorý označujeme epsilon)
- Neterminál S je "štartovací"
Použitie gramatiky na generovanie reťazcov
- Začneme so štartovacím neterminálom S
- V každom kroku prepíšeme najľavejší neterminál podľa niektorého pravidla
- Skončíme, keď nezostanú žiadne neterminály
- Príklad: S->aSb->aaSbb->aaaSbbb->epsilon
- Aké všetky slová vie táto gramatika generovať?
- V tvare aa...abb...b s rovnakým počtom á-čok a b-čiek (informatici píšu
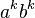 )
)
- V tvare aa...abb...b s rovnakým počtom á-čok a b-čiek (informatici píšu
Cvičenia
- Zostavte gramatiku na slova typu aa..abb..b kde acok je rovnako alebo viac ako bcok,
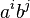 pre
pre 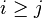
- S->aSb|aS|epsilon
- Zostavte gramatiku pre slova toho isteho typu, kde acok je viac ako bcok, t.j. i>j
- S->aSb|aT T->aT|epsilon (alebo S->aSb|aS|a)
- Zostavte gramatiku pre dobre uzatvorkovane vyrazy zo zatvoriek (,),[,]. Napr. [()()([])] je dobre uzatvorkovany, ale [(]) nie je.
- S->SS|(S)|[S]|epsilon
- priklad odvedenia v tejto gramatike: S->[S]->[SS]->[SSS]->[(S)SS]->[()SS]->[()(S)S]->[()()S]->[()()(S)]->[()()([S])]->[()()([])]
Parsovanie retazca pomocou gramatiky: urcit, ako mohol byt retazec vygenerovany pomocou pravidiel
- Gramatika pre dobre uzatvrokovane vyrazy nam pomoze urcit, ktora zatvorka patri ku ktorej: tie, ktore boli vygenerovane v jednom kroku
Dalsie cvicenia
- Zostavte gramatiku na DNA palindromy, t.j. sekvencie, ktore zozadu po skomplementovani baz daju to iste, ako napr. GATC
- S->gSc|cSg|aSt|tSa|epsilon
- Vlasenky RNA s lubovolne dlhou sparovanou castou a 3 nesparovanymi nukleotidmi v strede
- S->gSc|cSg|aSu|uSa|aaa|aac|aag|aau|...|uuu
- Tazsi priklad: Zostavte gramatiku na slova s rovnakym poctom acok a bcok v lubovolnom poradi
- S->epsilon|aSbS|bSaS
- ako bude generovat aababbba?
- preco vie vygenerovat vsetky take retazce?